昨年『R で2ちゃんねるを読んでみた』という遊びをやりました。詳細は読んでいただければ良いのですが,「Shift_JIS で定義されない文字が含まれているとうまく文字列処理ができないので,バイナリのまま処理した」という旨のことをサラッと書きましたが,その詳細については深く触れていません。そこで,あまり需要はないのかもしれませんが,実際に R でバイナリ処理を行う方法を簡単に紹介しようと思います。ただし,今回は読むだけで,書き込み操作に関しては扱いません。
なお,本記事は R Advent Calendar 2011 の 4 つ目の記事にあたります。
お題
バイナリといってもいろいろと種類がありますが,今回は Applied Biosystems の Genetic Analyzer が出力する ab1 ファイル (ABIF 形式) から,シークエンサーが読み取った波形データを取り出すというテーマで解説しようと思います。
サンプルデータが Example Data Sets の "Sequencing, Resequencing" から入手できます。アクセス時に Select Your Country
と聞かれますが, United States を選択してください。 Japan を選択すると,日本のサイトに飛ばされて,データが入手できません。
用語解説
いくつか事前に知っておくべき用語 (主にコンピューター関連) をごく簡単に解説します。誤りがあったらごめんなさい。各用語のリンクは,より詳しく知りたい方のための参考リンクです。
- シークエンス (Sequence)
- Genetic Analyzer はキャピラリー電気泳動装置です。 DNA 断片を電気泳動すると,断片サイズが小さいほど速く,大きいほどゆっくり移動します。対象配列中の各塩基まで伸長した DNA 断片の 3' 末端にあたる塩基 (AGCT) をそれぞれ異なる蛍光色素で標識して電気泳動し,それをレーザーに当てて CCD で蛍光強度を読み取ることで,塩基配列を決定することができます。つまりシークエンサーが読み取った蛍光の種類と強さの時系列データがファイルに保存されており,これを読み出すのが本記事の目的です。
- 符号付数値表現 (Signed Number Representation)
- 整数のビット列の 1 つを正負の判別に割り当てた数値表現です。普通は整数表現に用いられます。符号付の表現があるということは,逆に符号なし (unsigned) の表現もあるのですが,本記事では用いられません。
- エンディアン (Endianness)
- 1 つのデータを表すには,ほとんどの場合 1 バイト (8 ビット) では足りず,複数のバイトからなるバイト列により表されます。このバイトの並べ方には,データの上位から並べる方法と下位から並べる方法がありますが[A],この並び順のことをエンディアン,前者をビッグエンディアン (big endian),後者をリトルエンディアン (little endian) と言います。
例えば, 整数の 48879 を 4 バイトで表すと 0x0000BEEF になります。ビッグエンディアンでは00 00 BE EFの順に,リトルエンディアンではEF BE 00 00の順に保存されていることになります。 - コネクション (Connection)
- 操作される実体へのプロセスからの経路を指します。ファイル等の操作される実体は本来 R プログラムの外側にあるので,コネクションをつないで R から操作できるようにします。
- シーク (Seek)
- データを読み込む際に,読み込みの位置を一気に移動することです。パソコンの音楽再生ソフトで普通に曲を再生すると先頭から終端まで順番に進みますが,コントローラーをいじると任意の時間にジャンプする事ができるのと同じです[B]。ファイルを読む場合も先頭から終端まで順番に読めれば十分の場合もありますが, ABIF ファイルを読む際はシークが必須です。
- シークは必要か
- エンディアン
- (非 ASCII コードの文字列を扱う場合) エンコーディング
R による実装
早速実装に取り掛かりましょう。ABIF の仕様を調べながら実装していきます。仕様書は少しボリュームが大きいですが,実際に必要な部分はそんなにありません。
なお,本記事では R の対話環境で実行することを前提に説明します。
ファイルを開く
何はともあれ,まずファイルへのコネクションを確立することから始まります。
connection <- file("sample.ab1", "rb")
file 関数で read binary ("rb") モードでファイルを開くことができます。典型的にはファイルパスを指定しますが URL も指定できます[C]。ディスク上のファイルを読み込みモードで開く場合にはまず起こらない事ですが,ウェブ上のファイルを開く場合等に,もし isSeekable(connection) が FALSE である場合は,以下のコラムを参照してください。問題がなければ読み飛ばして構いません。
[tip]
ABIF ファイルはディレクトリという単位に情報がまとめられていて,ディレクトリにファイル中のどこに実データが記録されているか知ることができます。したがってファイルのシークが必要になるのですが,コネクションでつないだファイルがウェブ上に存在する場合等,シークができないという状況があります。シークができないと読み込みは順方向にしか進めません。後ろに戻るためにはコネクションを開き直す必要があります。それは無駄なので,ファイルの内容すべてをバイト列として一気読みしてメモリ上に確保してしまいましょう。
bytes <- raw(0)
while (length(buffer <- readBin(connection, raw(), 0x8000)) > 0)
{
bytes <- c(bytes, buffer)
}
close(connection)
ベクトルに読み込んだので,ベクトルのまま処理しても良いのですが,シーケンシャルな処理を行うにはコネクション経由の方が都合が良いです。幸いバイト列のためのコネクションを作成するための関数 rawConnection が用意されています。
connection <- rawConnection(bytes)
メモリ上のベクトルにコネクションを張っているので当然 isSeekable(connection) が TRUE になります。
なお,この方法はメモリ上にファイルの内容をすべて読み込むため,適切なメモリサイズが必要になります。 ABIF ファイルの場合は数百 KB 程度なので,まず問題にはならないでしょう。
[/tip]
ヘッダを読む
コネクションの準備ができたらバイナリを読み始めます。先頭の 128 バイトはヘッダで,ファイルの情報等が記述されています。
なお,操作を誤った場合は seek で戻る事ができます。
[tip]
コネクション経由の操作はシーケンシャル (順方向の一方向操作) になります。 readBin や readChar を実行すると seek 以外の操作で戻る事はできません。もし誤って読み込みすぎた場合は,以下の様に戻る事ができます。
## 現在の位置から 12 バイト戻る。 seek(connection, -12L, "current")
もし現在の位置がわからなくなってしまった場合,ファイルの先頭に戻る事もできます。
seek(connection, 0L, "start")
今回はシークできる前提なので必要はないはずですが,もしシークができない場合は,コネクションを一旦閉じ,再確立するとファイルの先頭から開く事ができます。
close(connection)
connection <- file("...")
[/tip]
シグネチャ
先頭の 4 バイトはシグネチャ,つまりそのファイルが本当に ABIF ファイルであるかどうかを示す文字列です。具体的には ABIF の 4 文字が入っています。
readString <- function(con, length=1L) readChar(con, length, useBytes=TRUE) fileSignature <- readString(connection, 4L)
このコードは, connection から 4 バイトを 4 つの文字として読む,という意味になります。コネクションの種類によっては useBytes を TRUE と明示しないと警告が出ます。なのでそれを避ける意味で readString というラッパーを作成しました。
[note]
ここで fileSignature == "ABIF" が FALSE ならば,このファイルは ABIF ファイルではないという判断ができます。しかし逆に TRUE だからといってファイルが破損している可能性もあります。
[/note]
バージョン番号
続く 2 バイトはバージョン番号を表す整数です。 ABIF ファイルでは,すべての整数はビッグエンディアンで記録されています。
readSInt16 <- function(con, n=1L) readBin(con, integer(), n=n, size=2L, signed=TRUE, endian="big") versionNumber <- readSInt16(connection)
readSInt16 関数は 16 ビットの符号付整数を読む関数です。中身の readBin はコネクションからバイトを読み込む関数で,読み込む結果の型,読み込む要素の個数,読み込む要素のサイズ,符号,エンディアンが指定できます。つまり,サイズが 2 バイトでビッグエンディアン形式の符号付数値表現の整数を n 個読み込め,という意味です。 readBin で読み込む型は整数の他にもバイトや実数といった R の基本データ型が選べます。詳しくはヘルプを見ましょう。
先ほど定義した readString もそうですが,引数が多いとミスをしやすいので,出来るだけ簡単な関数を作るとバグを見つけやすくなります。以下でも必要に応じてそのような関数を定義していきます。
ファイル構造
次の 28 バイトはファイル構造を示すディレクトリです。上で少し触れましたが, ABIF ファイルのデータはディレクトリと呼ばれるデータのまとまりです。すべてのディレクトリは 28 バイトからなる以下のようなバイト列です。
| 種類 | データの型 | 説明 |
|---|---|---|
| 名前 | 32 ビット符号付整数 | 定義上は整数だが,実際は文字列 |
| タグ番号 | 32 ビット符号付整数 | 同一名のディレクトリを区別するための番号 |
| 要素の型 | 16 ビット符号付整数 | 実データの型を示す整数 |
| 要素のサイズ | 16 ビット符号付整数 | 実データの 1 要素あたりのサイズ |
| 要素数 | 32 ビット符号付整数 | 実データの要素数 |
| データサイズ | 32 ビット符号付整数 | 実データのサイズ |
| オフセット | 32 ビット符号付整数 | 実データのファイル内での位置 (先頭から何バイト目から始まるか) ただしデータサイズが 4 バイト以下の場合は実データそのものが格納される |
| データハンドル | 32 ビット符号付整数 | 予約領域 (使用されない) |
ややこしいですね。特に 7 つ目の但し書きが面倒ですが,とりあえず無視して,ディレクトリを読む関数を作成しましょう。
readSInt32 <- function(con, n=1L)
readBin(con, integer(), n=n, size=4L, signed=TRUE, endian="big")
readDirectory <- function(con) {
name <- readString(con, 4L)
number <- readSInt32(con)
elementtype <- readSInt16(con)
elementsize <- readSInt16(con)
numelements <- readSInt32(con)
datasize <- readSInt32(con)
dataoffset <- readSInt32(con)
datahandle <- readSInt32(con)
list(
Name=name,
TagNumber=number,
ElementType=elementtype,
ElementSize=elementsize,
NumberOfElements=numelements,
DataSize=datasize,
DataOffset=dataoffset,
DataHandle=datahandle # Not used.
)
}
fileStructure <- readDirectory(connection)
定義に従うなら name は readSInt32 で読むべきですが,仕様書に ASCII 文字列として扱うべきと書いてあるので,そのようにしています。実際これで問題ないと思います。
正しく読み込めば, Name, TagNumber, ElementType, ElementSize にはそれぞれ "tdir", 1, 1023, 28 が入っているはずです。 28 というのはディレクトリのサイズを指します。 DataOffset の位置に, NumberOfElements 個のディレクトリが並んでいることがわかります。
未使用領域
ファイル構造まで読んでしまえば,実はもうシークしてしまって良いのですが,練習として最後までヘッダを読みましょう。と言ってもヘッダの残りの 94 バイトは, 47 個の 2 バイト整数です。すべて 0 です。
unusedSpace <- readSInt16(connection, n=47L)
データの読み込み
ディレクトリの取得
ファイル構造で取得したディレクトリの位置に移動して,実際のデータの取得の準備に取り掛かります。
seek(connection, fileStructure$DataOffset)
今回取得したいのはクロマトグラムのみなので,取得したいデータに関係するディレクトリは FWO_ と DATA の 2 つです。 DATA ディレクトリは複数あって, TagNumber が 1–4 のディレクトリにそれぞれ読み取った蛍光強度が記録されています。 1–4 がどの塩基に対応しているかを示すのが FWO_ ディレクトリの要素です。
fluorescence <- data.frame()
for (i in seq(1, fileStructure$NumberOfElements)) {
directory <- readDirectory(connection)
if (directory$Name == "FWO_") {
baseOrder <- directory
} else if (directory$Name == "DATA" &&
(1 <= directory$TagNumber && directory$TagNumber <= 4)) {
fluorescence <- rbind(fluorescence, directory)
}
}
蛍光強度のデータは 4 つあるので,後で参照するためにデータフレームにまとめておきます。
塩基の順番を取得
先にも述べた通り, baseOrder には塩基の順番が記されています。さて, baseOrder$DataOffset の値を見ると, 1195463747 の様な大きな値になっています。ファイルサイズよりずっと大きな値です。この位置にシークしても,ファイルの外を見る事になってしまいます。これはどういうことでしょうか。
ここでディレクトリの仕様を思い出してみると,データオフセットは,データサイズが 4 以下の場合は実データそのものが格納されるというマジキチ仕様でした。確認すると baseOrder$DataSize は確かに 4 で 4 以下です。つまり baseOrder$DataOffset はオフセットではなくデータです。
データの型は baseOrder$ElementType が 2 なので文字で,要素数は baseOrder$NumberOfElements が 4 なので 4 要素であることがわかります。データサイズは,データ型が 1 であることからも baseOrder$ElementSize が 1 であることからも 1 バイトです。
さて,既に DataOffset を整数として読み込んでしまったので,これを文字として読み直す必要があります。普通なら readDirectory を修正するところですが,ここではその場しのぎ的な処理を行います。ちゃんとした処理を行いたい場合は readDirectory を修正する方が良いのですが,それは最後の「おまけ」に。
#install.packages("bitops")
library(bitops)
rawOrder <- as.raw(bitAnd(bitShiftR(baseOrder$DataOffset, c(24, 16, 8, 0)), 0xFF))
rawOrder <- unlist(strsplit(rawToChar(rawOrder), ""))
fluorescence <- cbind(fluorescence, Base=rawOrder)
ABIF での整数はビッグエンディアンなので,整数の上位バイトから下位バイトへの順番が塩基の順番に相当します。ビット演算で整数を構成するバイト列を得たら rawToChar で文字列に変換し, strsplit(split="") で文字に分解します。 strsplit はリストで結果を返すので,データフレームに結合するために unlist でベクトルに変換しています。
電気泳動の結果を取得
いよいよ大詰めです。 fluorescence の中を見ればわかる通り,いずれの塩基も ElementType が 4 なので 16 ビット符号付整数の繰り返しです。
chromat <- list()
for (i in seq(1, nrow(fluorescence))) {
seek(connection, fluorescence[i, "DataOffset"])
response <- readSInt16(connection, fluorescence[i, "NumberOfElements"])
base <- as.character(fluorescence[i, "Base"])
chromat[[base]] <- response
}
後処理
最後に,開いたコネクションはキチンと閉じましょう[D]。
close(connection)
クロマトグラムのプロット
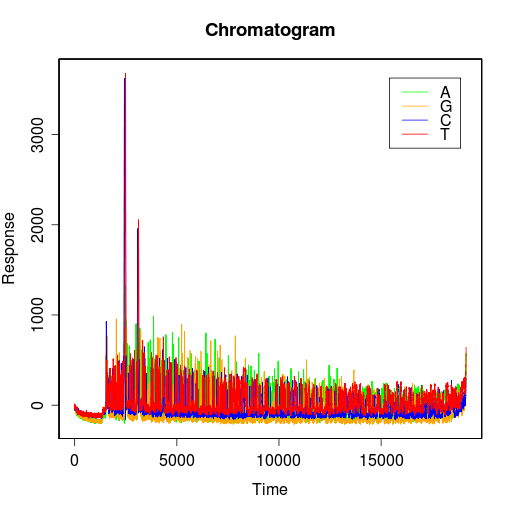
chromat$A, chromat$G, chromat$C, chromat$T はそれぞれの塩基の蛍光強度のベクトルになります。なので,普通に plot 関数でプロットできます。時系列データなので type="l" で表示してやれば良いでしょう。
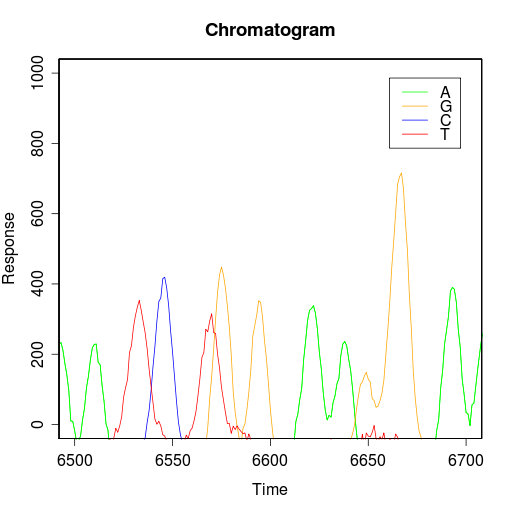
きちんと表示できましたね。これだと見辛いので,一部分を取り出してプロットしてみました。
この部分は AATCTGGAAGGAA となっていることが読み取れます。ちなみにクロマトグラムのピークを検出して塩基を判別する操作をベースコールと言います。著名なベースコールソフトウェアには Phred が挙げられます。ところで R で時系列データを持っているなら,自分でベースコールして塩基配列を決定するというのも難しくない気がしてきますね。ベースコールに関して私は真に驚くべき実装を見つけたが,この余白はそれを書くには狭すぎる。という事で,近いうちにベースコールの実装について書きたいと思います。
まとめ
バイナリを読む際に注意すべき主な点は,以下の通りです。
後はちゃんと仕様を調べるというのももちろん大事です。
さて, R でも簡単にバイナリを扱えることがわかっていただけたでしょうか。バイナリが扱えるようになると,扱えるデータの幅も広がるので,いろんなことに挑戦できるようになります。慣れないと難しいかもしれませんが,チャレンジしてみてください。
おまけ
ソースコードを Bitbucket にアップロードしています。本文中で手を抜いた readDirectory の処理は別の方法で実装したので bitops パッケージは不要です。プロット関数も書きました。お試し下さい。